
作者 | Jason Brownlee编译 | CDA数据分析师
使用实值数据(例如使用Pearson的相关系数)时,特征选择通常很简单,但是使用分类数据时可能会遇到挑战。
当目标变量也是分类的(例如分类预测建模)时,分类输入数据的两种最常用的特征选择方法是卡方统计和互信息统计。
在本教程中,您将发现如何使用分类输入数据执行特征选择。
完成本教程后,您将知道:
本教程分为三个部分:他们是:

作为本教程的基础,我们将使用自1980年代以来作为机器学习数据集而被广泛研究的所谓“ 乳腺癌 ”数据集。
该数据集将乳腺癌患者数据分类为癌症复发或无复发。有286个示例和9个输入变量。这是一个二进制分类问题。
天真的模型可以在此数据集上达到70%的精度。好的分数大约是76%+/- 3%。我们将针对该区域,但是请注意,本教程中的模型并未进行优化。它们旨在演示编码方案。
您可以下载数据集,然后将文件另存为“ breast-cancer.csv ”在当前工作目录中。
查看数据,我们可以看到所有九个输入变量都是分类的。
具体来说,所有变量都用引号引起来;有些是序数,有些不是。
'40-49','premeno','15-19','0-2','yes','3','right','left_up','no','recurrence-events' '50-59','ge40','15-19','0-2','no','1','right','central','no','no-recurrence-events' '50-59','ge40','35-39','0-2','no','2','left','left_low','no','recurrence-events' '40-49','premeno','35-39','0-2','yes','3','right','left_low','yes','no-recurrence-events' '40-49','premeno','30-34','3-5','yes','2','left','right_up','no','recurrence-events' ...
我们可以使用Pandas库将该数据集加载到内存中。
... # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values
加载后,我们可以将列分为输入(X)和输出以进行建模。
... # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1]
最后,我们可以将输入数据中的所有字段都强制为字符串,以防万一熊猫试图将某些字段自动映射为数字(确实如此)。
... # format all fields as string X = X.astype(str)
我们可以将所有这些结合到一个有用的功能中,以备后用。
# load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y
加载后,我们可以将数据分为训练集和测试集,以便我们可以拟合和评估学习模型。
我们将使用scikit-learn形式的traintestsplit()函数,并将67%的数据用于训练,将33%的数据用于测试。
... # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1)
将所有这些元素结合在一起,下面列出了加载,拆分和汇总原始分类数据集的完整示例。
# load and summarize the dataset from pandas import read_csv from sklearn.model_selection import train_test_split # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # summarize print('Train', X_train.shape, y_train.shape) print('Test', X_test.shape, y_test.shape)
运行示例将报告训练和测试集的输入和输出元素的大小。
我们可以看到,我们有191个示例用于培训,而95个用于测试。
Train (191, 9) (191, 1) Test (95, 9) (95, 1)
既然我们已经熟悉了数据集,那么让我们看一下如何对它进行编码以进行建模。
我们可以使用scikit-learn的OrdinalEncoder()将每个变量编码为整数。这是一个灵活的类,并且允许将类别的顺序指定为参数(如果已知这样的顺序)。
注意:我将作为练习来更新以下示例,以尝试为具有自然顺序的变量指定顺序,并查看其是否对模型性能产生影响。
对变量进行编码的最佳实践是使编码适合训练数据集,然后将其应用于训练和测试数据集。

下面名为prepare_inputs()的函数获取火车和测试集的输入数据,并使用序数编码对其进行编码。
# prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc
我们还需要准备目标变量。
这是一个二进制分类问题,因此我们需要将两个类标签映射到0和1。这是一种序数编码,而scikit-learn提供了专门为此目的设计的LabelEncoder类。尽管LabelEncoder设计用于编码单个变量,但我们可以轻松使用OrdinalEncoder并获得相同的结果。
所述prepare_targets()函数整数编码的训练集和测试集的输出数据。
# prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc
我们可以调用这些函数来准备我们的数据。
... # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
综上所述,下面列出了加载和编码乳腺癌分类数据集的输入和输出变量的完整示例。
# example of loading and preparing the breast cancer dataset from pandas import read_csv from sklearn.model_selection import train_test_split from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test)
现在我们已经加载并准备了乳腺癌数据集,我们可以探索特征选择。
有两种流行的特征选择技术,可用于分类输入数据和分类(类)目标变量。
他们是:
让我们依次仔细研究每个对象。
皮尔逊的卡方统计假设检验是分类变量之间独立性检验的一个示例。
您可以在教程中了解有关此统计测试的更多信息:
该测试的结果可用于特征选择,其中可以从数据集中删除与目标变量无关的那些特征。
scikit-learn机器库在chi2()函数中提供了卡方检验的实现。此功能可用于特征选择策略中,例如通过SelectKBest类选择前k个最相关的特征(最大值)。
例如,我们可以定义SelectKBest类以使用chi2 ()函数并选择所有功能,然后转换训练序列和测试集。
... fs = SelectKBest(score_func=chi2, k='all') fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test)
然后,我们可以打印每个变量的分数(越大越好),并将每个变量的分数绘制为条形图,以了解应该选择多少个特征。
... # what are scores for the features for i in range(len(fs.scores_)): print('Feature %d: %f' % (i, fs.scores_[i])) # plot the scores pyplot.bar([i for i in range(len(fs.scores_))], fs.scores_) pyplot.show()
将其与上一节中乳腺癌数据集的数据准备结合在一起,下面列出了完整的示例。
# example of chi squared feature selection for categorical data from pandas import read_csv from sklearn.model_selection import train_test_split from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import chi2 from matplotlib import pyplot # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=chi2, k='all') fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs, fs # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test) # feature selection X_train_fs, X_test_fs, fs = select_features(X_train_enc, y_train_enc, X_test_enc) # what are scores for the features for i in range(len(fs.scores_)): print('Feature %d: %f' % (i, fs.scores_[i])) # plot the scores pyplot.bar([i for i in range(len(fs.scores_))], fs.scores_) pyplot.show()

首先运行示例将打印为每个输入要素和目标变量计算的分数。
注意:您的具体结果可能会有所不同。尝试运行该示例几次。
在这种情况下,我们可以看到分数很小,仅凭数字很难知道哪个功能更相关。
也许功能3、4、5和8最相关。
Feature 0: 0.472553 Feature 1: 0.029193 Feature 2: 2.137658 Feature 3: 29.381059 Feature 4: 8.222601 Feature 5: 8.100183 Feature 6: 1.273822 Feature 7: 0.950682 Feature 8: 3.699989
创建每个输入要素的要素重要性得分的条形图。
这清楚地表明,特征3可能是最相关的(根据卡方),并且九个输入特征中的四个也许是最相关的。
在配置SelectKBest来选择这前四个功能时,我们可以设置k = 4 。
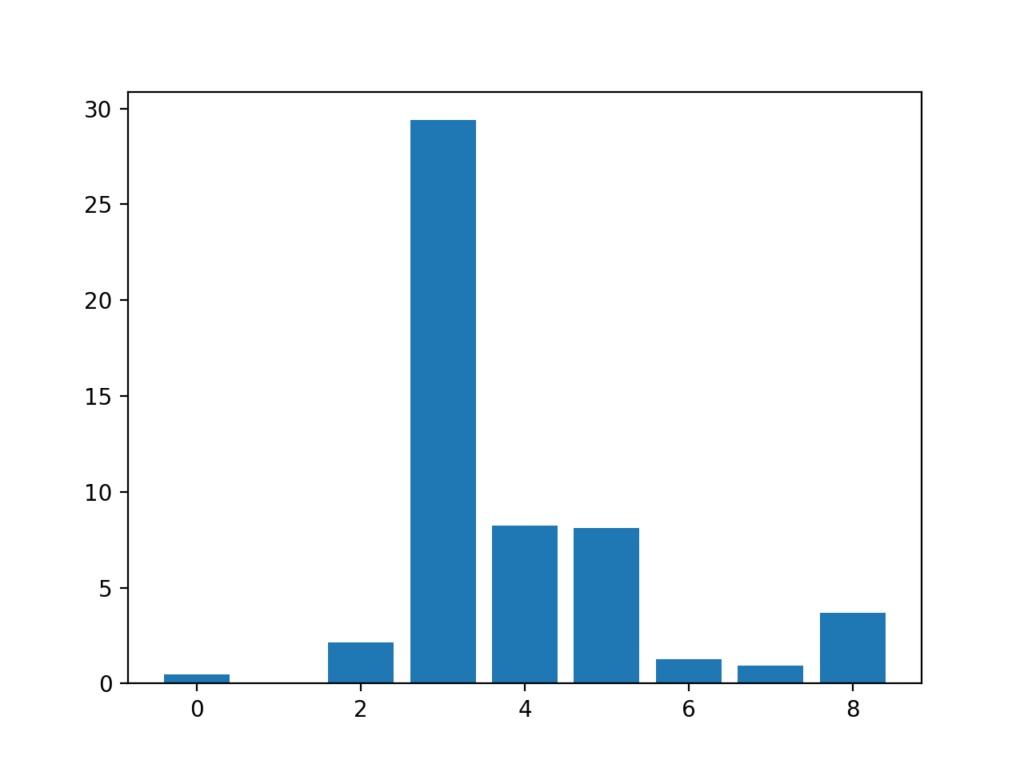
输入要素的条形图(x)vs Chi-Squared要素重要性(y)
来自信息理论领域的互信息是信息增益(通常用于决策树的构建)在特征选择中的应用。
在两个变量之间计算互信息,并在给定另一个变量的已知值的情况下测量一个变量的不确定性降低。
您可以在以下教程中了解有关相互信息的更多信息。
scikit-learn机器学习库通过commoninfoclassif()函数提供了用于信息选择的互信息实现。
像chi2()一样,它可以用于SelectKBest特征选择策略(和其他策略)中。
# feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=mutual_info_classif, k='all') fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs, fs
我们可以使用关于乳腺癌组的相互信息来进行特征选择,并像上一节中那样打印和绘制分数(越大越好)。
下面列出了使用互信息进行分类特征选择的完整示例。
# example of mutual information feature selection for categorical data from pandas import read_csv from sklearn.model_selection import train_test_split from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import mutual_info_classif from matplotlib import pyplot # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=mutual_info_classif, k='all') fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs, fs # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test) # feature selection X_train_fs, X_test_fs, fs = select_features(X_train_enc, y_train_enc, X_test_enc) # what are scores for the features for i in range(len(fs.scores_)): print('Feature %d: %f' % (i, fs.scores_[i])) # plot the scores pyplot.bar([i for i in range(len(fs.scores_))], fs.scores_) pyplot.show()
首先运行示例将打印为每个输入要素和目标变量计算的分数。
注意:您的具体结果可能会有所不同。尝试运行该示例几次。
在这种情况下,我们可以看到某些功能的得分很低,表明也许可以将其删除。
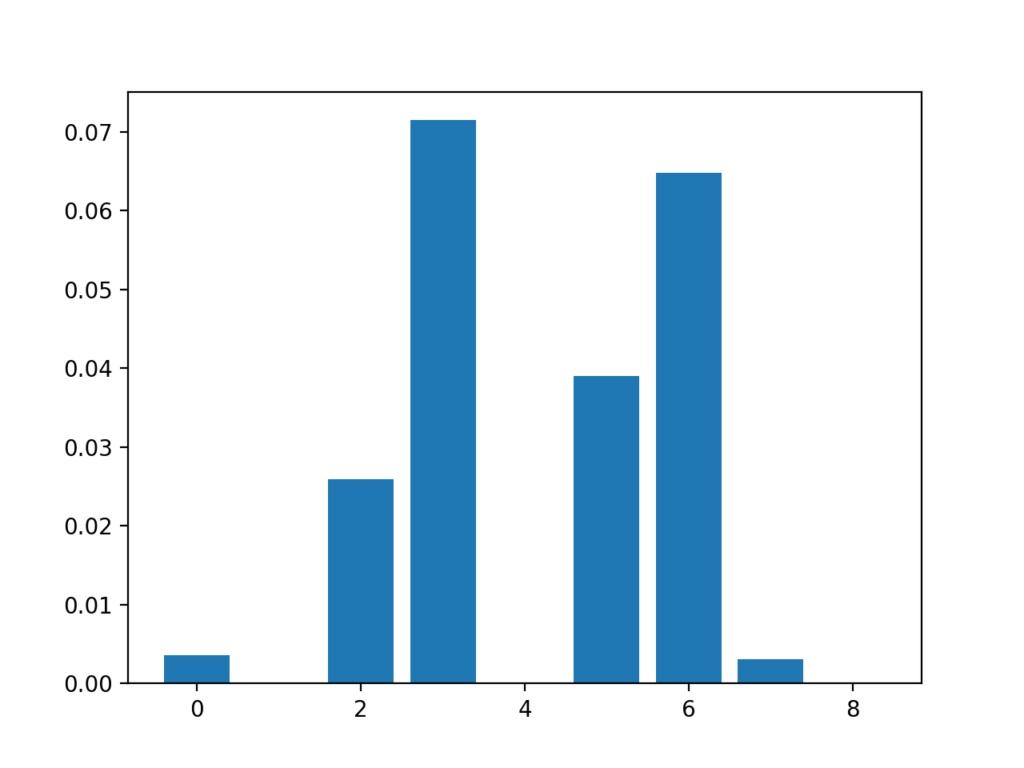
也许功能3、6、2和5最相关。
Feature 0: 0.003588 Feature 1: 0.000000 Feature 2: 0.025934 Feature 3: 0.071461 Feature 4: 0.000000 Feature 5: 0.038973 Feature 6: 0.064759 Feature 7: 0.003068 Feature 8: 0.000000
创建每个输入要素的要素重要性得分的条形图。
重要的是,促进了特征的不同混合。
既然我们知道如何针对分类预测建模问题对分类数据执行特征选择,那么我们可以尝试使用选定的特征开发模型并比较结果。
有许多不同的技术可用来对特征评分和根据分数选择特征。您怎么知道要使用哪个?
一种可靠的方法是使用不同的特征选择方法(和特征数量)评估模型,然后选择能够产生最佳性能的模型的方法。
在本节中,我们将评估具有所有要素的Logistic回归模型,并将其与通过卡方选择的要素和通过互信息选择的要素构建的模型进行比较。
逻辑回归是测试特征选择方法的良好模型,因为如果从模型中删除了不相关的特征,则逻辑回归性能会更好。
第一步,我们将使用所有可用功能评估LogisticRegression模型。
该模型适合训练数据集,并在测试数据集上进行评估。
下面列出了完整的示例。
# evaluation of a model using all input features from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder from sklearn.model_selection import train_test_split from sklearn.linear_model import LogisticRegression from sklearn.metrics import accuracy_score # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test) # fit the model model = LogisticRegression(solver='lbfgs') model.fit(X_train_enc, y_train_enc) # evaluate the model yhat = model.predict(X_test_enc) # evaluate predictions accuracy = accuracy_score(y_test_enc, yhat) print('Accuracy: %.2f' % (accuracy*100))
运行示例将在训练数据集上打印模型的准确性。
注意:根据学习算法的随机性,您的特定结果可能会有所不同。尝试运行该示例几次。
在这种情况下,我们可以看到该模型实现了约75%的分类精度。
我们宁愿使用能够实现比此更好或更高的分类精度的功能子集。
Accuracy: 75.79

下面的select_features()函数已更新以实现此目的。
# feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=chi2, k=4) fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs
下面列出了使用这种特征选择方法评估逻辑回归模型拟合和对数据进行评估的完整示例。
# evaluation of a model fit using chi squared input features from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import chi2 from sklearn.model_selection import train_test_split from sklearn.linear_model import LogisticRegression from sklearn.metrics import accuracy_score # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=chi2, k=4) fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test) # feature selection X_train_fs, X_test_fs = select_features(X_train_enc, y_train_enc, X_test_enc) # fit the model model = LogisticRegression(solver='lbfgs') model.fit(X_train_fs, y_train_enc) # evaluate the model yhat = model.predict(X_test_fs) # evaluate predictions accuracy = accuracy_score(y_test_enc, yhat) print('Accuracy: %.2f' % (accuracy*100))
运行示例将报告使用卡方统计量选择的九个输入要素中只有四个要素的模型性能。
注意:根据学习算法的随机性,您的特定结果可能会有所不同。尝试运行该示例几次。
在这种情况下,我们看到该模型的准确度约为74%,性能略有下降。
实际上,某些已删除的功能可能会直接增加价值,或者与所选功能一致。
在这个阶段,我们可能更喜欢使用所有输入功能。
Accuracy: 74.74
我们可以重复实验,并使用相互信息统计量选择前四个功能。
下面列出了实现此目的的select_features()函数的更新版本。
# feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=mutual_info_classif, k=4) fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs
下面列出了使用互信息进行特征选择以拟合逻辑回归模型的完整示例。
# evaluation of a model fit using mutual information input features from pandas import read_csv from sklearn.preprocessing import LabelEncoder from sklearn.preprocessing import OrdinalEncoder from sklearn.feature_selection import SelectKBest from sklearn.feature_selection import mutual_info_classif from sklearn.model_selection import train_test_split from sklearn.linear_model import LogisticRegression from sklearn.metrics import accuracy_score # load the dataset def load_dataset(filename): # load the dataset as a pandas DataFrame data = read_csv(filename, header=None) # retrieve numpy array dataset = data.values # split into input (X) and output (y) variables X = dataset[:, :-1] y = dataset[:,-1] # format all fields as string X = X.astype(str) return X, y # prepare input data def prepare_inputs(X_train, X_test): oe = OrdinalEncoder() oe.fit(X_train) X_train_enc = oe.transform(X_train) X_test_enc = oe.transform(X_test) return X_train_enc, X_test_enc # prepare target def prepare_targets(y_train, y_test): le = LabelEncoder() le.fit(y_train) y_train_enc = le.transform(y_train) y_test_enc = le.transform(y_test) return y_train_enc, y_test_enc # feature selection def select_features(X_train, y_train, X_test): fs = SelectKBest(score_func=mutual_info_classif, k=4) fs.fit(X_train, y_train) X_train_fs = fs.transform(X_train) X_test_fs = fs.transform(X_test) return X_train_fs, X_test_fs # load the dataset X, y = load_dataset('breast-cancer.csv') # split into train and test sets X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.33, random_state=1) # prepare input data X_train_enc, X_test_enc = prepare_inputs(X_train, X_test) # prepare output data y_train_enc, y_test_enc = prepare_targets(y_train, y_test) # feature selection X_train_fs, X_test_fs = select_features(X_train_enc, y_train_enc, X_test_enc) # fit the model model = LogisticRegression(solver='lbfgs') model.fit(X_train_fs, y_train_enc) # evaluate the model yhat = model.predict(X_test_fs) # evaluate predictions accuracy = accuracy_score(y_test_enc, yhat) print('Accuracy: %.2f' % (accuracy*100))
运行示例使模型适合于使用互信息选择的前四个精选功能。
注意:根据学习算法的随机性,您的特定结果可能会有所不同。尝试运行该示例几次。
在这种情况下,我们可以看到分类精度小幅提升至76%。
为了确保效果是真实的,最好将每个实验重复多次并比较平均效果。探索使用k倍交叉验证而不是简单的训练/测试拆分也是一个好主意。

数据分析咨询请扫描二维码
CDA数据分析师在中国航信高科技产业园进行了面向测试度量的数据分析培训课程,培训人数近2 ...
2024-05-01CDA数据分析师走进深圳迈瑞生物医疗电子股份有限公司,在迈瑞总部展开了为期两天的培训,本次课程参训人员线上及线下近百人, ...
2024-05-01CDA数据分析师在合肥市对合肥阳光新能源科技有限公司开展了为期8天的企业内训。 合肥阳光新能源科技 ...
2024-05-01CDA数据分析师走进海尔大学,进行了《数据治理与数据中台建设的道与术》专题培训,培训现场爆满,近百人参加了此次培训。 ...
2024-05-01在中国银行苏州分行培训中心开始数据分析师培训,此次培训课程共10天内容,包括Excel、MySQL、概率论与数理统计、SPSS等内容, ...
2024-05-01从实际的业务需求出发,结合行业的典型应用特点,围绕实际的商业问题,探讨数据挖掘、机器学习模型在金融领域的应用,包括获客、信用评分、细分画像、交叉销售、反欺诈、违规识别、时序预测、运筹优化、流程挖掘九个方面,形成 ...
2024-05-01本次培训课程为线上+线下的模式,由于学员编程能力不一、部分学员没有编程基础,故提供统计学、python基 ...
2024-05-01华夏银行信用卡中心-机器学习培训 1、课程亮点 取材于业界一流企业和顶级咨询公司的行业实践;已经被证明是人人 ...
2024-05-01主 题:数据中台建设及数据分析应用主题分享 1. 数据中台市场洞察 2. 主流数据中台产品比较 3. 某企业数据中 ...
2024-05-01围绕“数据驱动”战略,全力打造我行 300 人数字化人才梯队,着力培养数字化管理人才、大数据专业团队 ...
2024-05-01在当今数据驱动的商业环境中,数据分析成为了企业决策的重要依据。通过对大量数据的收集、处理和分析,企业能够更好地理解市场 ...
2024-04-29在人工智能(AI)的世界里,提示词(Prompt)是一种强大的工具,它能够引导AI按照用户的需求产生特定的输出。本文将深入探讨AI ...
2024-04-29CDA立足未来职场,拓展前沿视野——对外经贸大学保险学院举办“三全育人大讲堂”分享行业最新动态。 ...
2024-04-294月2日,CDA数据分析师创始发起人兼协会理事长赵坚毅博士受邀在浙江万里学院举办了一场以“数字化能力在职场中的作用” ...
2024-04-29随机森林(Random Forests)现在机器学习中比较火的一个算法,是一种基于Bagging的集成学习方法,能够很好地处理分类和回归的问 ...
2022-12-23方差分析是数据分析中常用的一种统计分析方法,接下来让我们简单了解一下方差分析的基本思想和原理吧。 方差分析(Analysis ...
2022-12-23来源:关于数据分析与可视化 关于streamlit-aggrid 数据排序 表格样式的调整 数据 ...
2022-08-03作者:麦叔 定义 「把上面晦涩的概念汇成一句话就是:」 ❝ 回调函数就是一个被作为参 ...
2022-08-03现今,高学历人群日益增多,物以稀为贵的高学历光环淡去。无论本科生还是研究生,甚至博士生,求职竞争力都大不如前,就业压力越来越大。
2022-06-01某家企业10个人面试,有9个本科生……如何脱颖而出,除得体的举止和良好的沟通力外,证书成重要筹码,这也是很多人考证的关键所在。
2022-04-14